Основной вычислительной задачей, поставленной перед Центром высокопроизводительных вычислений, является сборка (ассемблирование) генома сибирской лиственицы и сибирской кедровой сосны - основных видов хвойных бореальных лесов Сибири, являющихся лесным резервом России. Геномы хвойных уникальны по размеру (в 4-7 раз больше генома человека) и очень сложны для изучения. По мере развития планируются генетические исследования геномов других хозяйственно-важных видов растений и животных, а также, медико-генетические исследования генома человека.
На базе Центра геномных исследований и Центра высокопроизводительных вычислений СФУ выполняются работы по созданию сервиса для решения широкого круга задач в области геномных исследований и клинической геномики.
Сервис предполагает включение в него специализированного биохимического оборудования, высокопроизводительного оборудования с уникальными характеристиками, специализированного проприетарного ПО и ПО с открытым кодом (Рисунок 1), что позволит обеспечить биологов и медиков возможностями реконструировать и анализировать последовательности полного генома человека, животных, растений.
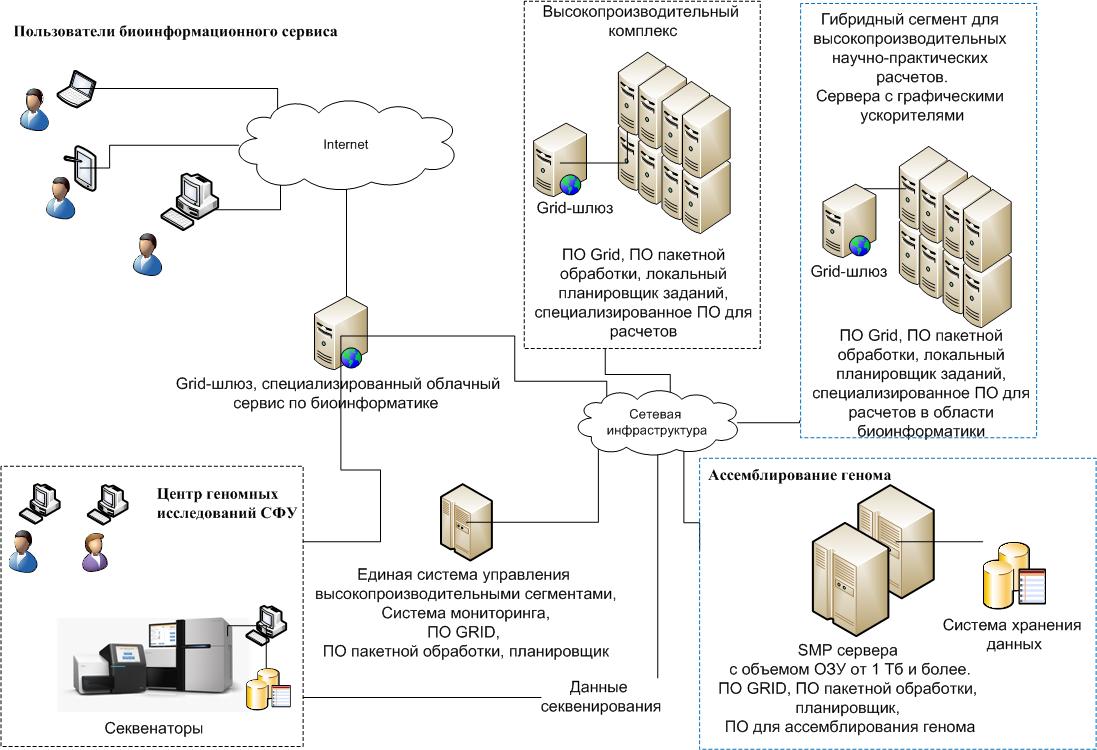
Рисунок 1. Схема организации сервиса геномных исследований
Реализация сервиса предполагает:
• развертывание комплекса оборудования для секвенирования генома;
• создание специализированного высокопроизводительного комплекса с объемом оперативной памяти от 2 до 3 Тб, с набором специализированного программного обеспечения с открытым кодом и проприетарным ПО, реализующего широкий диапазон задач биоинформатики. Комплекс обеспечит эффективную обработку и хранение данных, производимых Центром геномных исследований СФУ.
• создание и адаптацию биоинформатических алгоритмов для гибридных кластерных систем.
• создание облачного сервиса, предоставляющего интерфейс обработки задач биоинформатики на распределенных суперкомпьютерных ресурсах.
Уже внедрено и работает следующее оборудование
в том числе уникальный, для региона, специализированный вычислительный комплекс с суммарным объемом ОЗУ больше 3 Тб и с возможностью расширения памяти до 12 Тб.
В состав комплекса входят:
96 ядерный SMP сервер IBM x3950 X6 с объемом ОЗУ 3 ТБ;

Гибридный счетный сервер IBM dx360 M4 c двумя GPU NVIDIA Tesla K20 обеспечивающий суммарную пиковую производительность для вычислений одинарной точности с плавающей точкой до 7 Tflops;

Подсистема хранения данный IBM Storwize V3700 объемом 72 Тб.

Комплекс работает под управлением ОС Centos7, установлена параллельная файловая система IBM GPFS, система мониторинга Ganglia, система пакетной обработки Torque.
Развернуто специализированное ПО для обработки геномной информации в том числе геномные ассемблеры CLC Assembler Cell, ABySS, MaSuRCA, SPAdes, Platanus, ПО Blast, BWA.




Результаты представлены в следующих публикациях:
Putintseva Yu.A., E.I. Bondar, E.P. Simonov, V.V. Sharov, N.V. Oreshkova, D.A. Kuzmin, Yu.M. Konstantinov, V.N. Shmakov, V.I. Belkov, M.S. Sadovsky, O. Keech, K.V. Krutovsky 2020 Siberian larch (Larix sibirica Ledeb.) mitochondrial genome assembled using both short and long nucleotide sequence reads is currently the largest known mitogenome. BMC Genomics 21: 654 https://doi.org/10.1186/s12864-020-07061-4 (IF = 3.594; Q2)
Kuzmin, D. A., S. I. Feranchuk, V. V. Sharov, A. N. Cybin, S. V. Makolov, Y. A. Putintseva, N. V. Oreshkova, K. V. Krutovsky, 2019 Stepwise large genome assembly approach: A case of Siberian larch (Larix sibirica Ledeb.). BMC Bioinformatics 20(Suppl. 1) doi: 10.1186/s12859-018-2570-y (IF = 2.213; Q1)
Sadovsky M., Kobets V., Khodos G., Kuzmin D., Sharov V. (2019) Reads in NGS Are Distributed over a Sequence Very Inhomogeneously. In: Rojas I., Valenzuela O., Rojas F., Ortuño F. (eds) Bioinformatics and Biomedical Engineering. IWBBIO 2019. Lecture Notes in Computer Science, vol 11465. Springer, Cham DOI https://doi.org/10.1007/978-3-030-17938-0_25 (Scopus)
Bondar, A. Kirichenko, V. Sharov, Yu. Putintceva, N. Oreshkova, S. Feranchuk, Yu. Konstantinov, V. Shmakov, V. Belkov, D. Kuzmin, S. Sadovsky, K. Krutovsky Sequencing and Assembly of Mitochondrial Genomes in Three Conifer Species Larix sibirica, Pinus sibirica and Pinus sylvestris // The Multiconference BGRS\SB-2018, 20-25.08 Novosibirsk. p. 153.
A. Kolesnikova, Y. Putintseva, S. Jain, N. Oreshkova, I. Pavlov, V. Sharov, D. Kuzmin, S. Makolov, K. Krutovsky Rapid Evolution of Mitochondrial Genomes in Three Closely-Related Armillaria Species // The Multiconference BGRS\SB-2018, 20-25.08 Novosibirsk. p. 54.
Yu. Putintseva, V. Sharov, D. Kuzmin, N. Oreshkova, S. Feranchuk, V. Biryukov, S. Novikova, K. Miroshnikova, S. Makolov, M. Sadovsky, K. Krutovsky Genomes of Three Conifer Species: Larix sibirica, Pinus sibirica and Pinus sylvestris // The Multiconference BGRS\SB-2018, 20-25.08 Novosibirsk. p. 71.
Oreshkova N. V., Putintseva Y. A.,Sharov V. V. Kuzmin D. A., Krutovsky K. V. Development of microsatellite genetic markers in Siberian larch (Larix sibirica Ledeb.) based on the de novo whole genome sequencing // Russian Journal of Genetics, 2017, Т.53, № 11. –С. 1194-1199, ISSN 1022-7954. (Web of Science, SCOPUS)
Орешкова Н.В., Путинцева Ю.А., Шаров В.В., Кузьмин Д.А., Крутовский К.В. Разработка микросателлитных маркеров лиственницы сибирской (larix sibirica ledeb.) На основе полногеномного de novo секвенирования //ГЕНЕТИКА, 2017. -Т.53, №11., г. Москва. - С. 1278-1284. ISSN: 0016-6758 (РИНЦ)
Yu. Putintseva, V. Sharov, D. Kuzmin, N. Oreshkova, S. Feranchuk, V. Biryukov, S. Novikova, K. Miroshnikova, S. Makolov, M. Sadovsky, K. Krutovsky Genomes of Three Conifer Species: Larix sibirica, Pinus sibirica and Pinus sylvestris // The Multiconference BGRS\SB-2018, 20-25.08 Novosibirsk. p. 71.
Bondar, A. Kirichenko, V. Sharov, Yu. Putintceva, N. Oreshkova, S. Feranchuk, Yu. Konstantinov, V. Shmakov, V. Belkov, D. Kuzmin, S. Sadovsky, K. Krutovsky Sequencing and Assembly of Mitochondrial Genomes in Three Conifer Species Larix sibirica, Pinus sibirica and Pinus sylvestris // The Multiconference BGRS\SB-2018, 20-25.08 Novosibirsk. p. 153.
Кириченко А. Д., СФУ; Бондар Е. И., СФУ; Шаров В.В. Сборка митохондриального генома сосны сибирской кедровой (Pinus sibirica Du Tour)// Международная научная студенческая конференция 2018, Новосибирск, 22 апреля 2018 г. – 27 апреля 2018 г.
Шаров В.В., Путинцева Ю.А., Кузьмин Д.А., Орешкова Н.В., Феранчук С.И., Цыбин А.Н., Маколов С.В., Крутовский К.В. 2017 Сборка и аннотирование генома лиственницы сибирской. Программа II Всероссийской конференции с международным участием «Высокопроизводительное секвенирование в геномике», 18–23 июня 2017 г., Новосибирск, РФ, С. 12
Шаров В.В., Путинцева Ю.А., Кузьмин Д.А., Орешкова Н.В., Феранчук С.И., Цыбин А.Н., Маколов С.В., Крутовский К.В. Сборка и аннотирование генома лиственницы сибирской // Acta Naturae, Всероссийская конференция с международным участием «Высокопроизводительное секвенирование в геномике», 2017. -Т. 9, №1., г. Новосибирск. -С. 63. (РИНЦ)
Феранчук С. И., Шаров В. В., Путинцева Ю. А., Кузьмин Д. А., Орешкова Н. В., Крутовский К. В. Функциональная аннотация генома лиственницы как подход к прояснению механизмов формирования важных генетических признаков // Материалы 5-ой Международной конференции-совещания "Сохранение лесных генетических ресурсов", 2017. -С. 225-228.
А.Н. Цыбин, В.В. Шаров, Ю.А. Путинцева, С.И. Феранчук, Д.А. Кузьмин «Параллельный алгоритм фильтрации повторов в данных NGS ILLUMINA» Доклады Академии Наук ВШ РФ, 2016 № 4 (33), сс. 99–110. doi: 10.17212/1727-2769-2016-4-99-110 (ВАК)
Oreshkova N.V., Putintseva Yu.A., Kuzmin D.A., Sharov V.V., Biryukov V.V., Makolov S.V., Deych K.O., Ibe A.A., Shilkina E.A., Krutovsky K.V. The whole de novo genome sequencing and assembly of Siberian larch (Larix sibirica Ledeb.) and Siberian pine (Pinus sibirica Du Tour.) // The 3rd International Conference «Plant genetics, genomics, bioinformatics and biotechnology» (PlantGen 2015) June 17-21, 2015. Novosibirsk, Russia, P. 37
Krutovsky K.V., Oreshkova N.V., Putintseva Yu.A., Kuzmin D.A., Sharov V.V., Biryukov V.V., Makolov S.V., Deych K.O., Bondar E.I., Ushakova O.A., Ibe A.A., Shilkina E.A. De novo sequencing of conifer megagenomes // The 3rd International Conference «Plant genetics, genomics, bioinformatics and biotechnology» (PlantGen 2015) June 17-21, 2015. Novosibirsk, Russia, P. 28.
Putintseva Yu.A., Sharov V.V., Kuzmin D.A., Makolov S.V., Oreshkova N.V., Krutovsky K.V. Challenges of assembling huge conifer genomes // The 3rd International Conference «Plant genetics, genomics, bioinformatics and biotechnology» (PlantGen 2015) June 17-21, 2015. Novosibirsk, Russia, P. 43.
Орешкова Н.В., Путинцева Ю.А., Кузмин Д.А., Шаров В.В.., Бирюков В.В., Дейч К.О., Ибе A.A., Шилкина E.A., Крутовский К.В. Секвенирование и сборка геномов лиственницы сибирской (Larix sibirica Ledeb.) и сосны кедровой сибирской (Pinus sibirica Du Tour) и предварительные данные анализа транскриптома // Материалы 4-го Международного совещания по сохранению лесных генетических ресурсов Сибири, Барнаул, 2015, С. 127-129.
Krutovsky K.V., Oreshkova N.V., Putintseva Yu.A., Pavlov I.N., Kuzmin D.A., Sharov V.V., Biryukov V.V., Makolov S.V., Deych K.O., Bondar E.I., Ushakova O.A., Ibe A.A., Shilkina E.A., Sadovsky M.G., Vaganov E.A. Pinus sibirica and Larix sibirica whole genome de novo sequencing// ProCoGen final open conference Promoting Conifer Genomic Resources 30th November – 2nd December 2015 Orléans, France, P. O-07